Originalmente publicado a 3 de Maio de 2017 e actualizado pela última vez a 24 de Setembro de 2020
Este post foi contribuído pelo blogger convidado, membro do Conselho Consultivo da Addgene, e cientista do Instituto Broad Institute, John Doench.
A tecnologiaCRISPR tornou mais fácil do que nunca tanto a engenharia de edições específicas de ADN como a realização de ecrãs funcionais para identificar genes envolvidos num fenótipo de interesse. Este post no blog discutirá as diferenças entre estas abordagens, e fornecerá actualizações sobre a melhor forma de conceber gRNAs. Pode também encontrar gRNAs validados para a sua próxima experiência em Addgene’s Validated gRNA Sequence Datatable. Uma discussão mais alargada destes assuntos pode ser encontrada em dois artigos de revisão recentes (Doench et al., 2017, e Hanna et al., 2020) e respectivas referências.
br>>p>
Nom nom. Eu amo gRNAs! BD de Maya Kostman.
Considerações importantes antes de começar uma experiência com CRISPR
O martelo, o quebra-cabeças, e a chave inglesa são todas grandes ferramentas, mas qual delas usa, claro, depende do que está a tentar fazer – não há nenhuma “melhor” ferramenta entre elas. Embora isto pareça óbvio, é importante lembrar que o mesmo é verdade ao conceber gRNAs para utilizar a tecnologia CRISPR – o “melhor” gRNA depende muito do que está a tentar fazer: eliminação de genes, uma edição de base específica, ou modulação da expressão genética.
 Localização e sequência são considerações importantes para conceber os seus gRNAs. Para indels, não é tão importante qual a localização no gene alvo, mas é importante que a sua sequência de gRNA seja concebida para ser altamente activa e reduzir os alvos. Para CRISPRa e CRISPRi, estas considerações são de importância aproximadamente igual (o alvo deve estar próximo do TSS mas pode preocupar-se menos com a optimização da sequência porque geralmente tem menos sequências por onde escolher). Finalmente, para o HDR, a localização é muito mais importante porque tem de ter como alvo ~30 nt da edição proposta, o que significa que há tão poucas gRNAs a escolher de entre essas preferências de sequência têm de ser largamente ignoradas.
Localização e sequência são considerações importantes para conceber os seus gRNAs. Para indels, não é tão importante qual a localização no gene alvo, mas é importante que a sua sequência de gRNA seja concebida para ser altamente activa e reduzir os alvos. Para CRISPRa e CRISPRi, estas considerações são de importância aproximadamente igual (o alvo deve estar próximo do TSS mas pode preocupar-se menos com a optimização da sequência porque geralmente tem menos sequências por onde escolher). Finalmente, para o HDR, a localização é muito mais importante porque tem de ter como alvo ~30 nt da edição proposta, o que significa que há tão poucas gRNAs a escolher de entre essas preferências de sequência têm de ser largamente ignoradas.
O martelo: Gene knockout por NHEJ
br>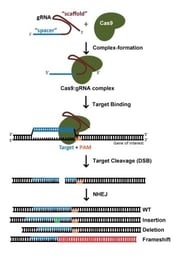 O knockout de genes com a tecnologia CRISPR é normalmente realizado por dsDNA breaks mediados por Cas9: após um corte, a natureza de junção final não-homológica (NHEJ), que é susceptível de erro, leva muitas vezes à geração de indels e, portanto, a mudanças de estrutura que perturbam a capacidade de codificação de proteínas de um locus. Ao utilizar S. pyogenes Cas9 (SpCas9), os potenciais locais alvo são ambos e , como é igualmente eficaz visar a vertente codificadora ou não codificadora do ADN. Como regra geral, evitamos locais alvo que codificam os aminoácidos perto do terminal N’ da proteína, a fim de mitigar a capacidade da célula de utilizar um ATG alternativo a jusante do códão de início anotado. Do mesmo modo, evitamos locais alvo que codificam para aminoácidos próximos do terminal C’ da proteína, para maximizar as hipóteses de criação de um alelo não funcional. Para um gene de 1 kilobase, uma vez que os potenciais locais alvo ocorrem ~1 em cada 8 nucleótidos, a restrição dos gRNAs a 5 – 65% da região de codificação da proteína ainda resultará em muitas dezenas de gRNAs à escolha. Com tantas possibilidades, escolher um gRNA com uma sequência optimizada é de primordial importância (mais sobre isto abaixo).
O knockout de genes com a tecnologia CRISPR é normalmente realizado por dsDNA breaks mediados por Cas9: após um corte, a natureza de junção final não-homológica (NHEJ), que é susceptível de erro, leva muitas vezes à geração de indels e, portanto, a mudanças de estrutura que perturbam a capacidade de codificação de proteínas de um locus. Ao utilizar S. pyogenes Cas9 (SpCas9), os potenciais locais alvo são ambos e , como é igualmente eficaz visar a vertente codificadora ou não codificadora do ADN. Como regra geral, evitamos locais alvo que codificam os aminoácidos perto do terminal N’ da proteína, a fim de mitigar a capacidade da célula de utilizar um ATG alternativo a jusante do códão de início anotado. Do mesmo modo, evitamos locais alvo que codificam para aminoácidos próximos do terminal C’ da proteína, para maximizar as hipóteses de criação de um alelo não funcional. Para um gene de 1 kilobase, uma vez que os potenciais locais alvo ocorrem ~1 em cada 8 nucleótidos, a restrição dos gRNAs a 5 – 65% da região de codificação da proteína ainda resultará em muitas dezenas de gRNAs à escolha. Com tantas possibilidades, escolher um gRNA com uma sequência optimizada é de primordial importância (mais sobre isto abaixo).
O quebra-cabeças: Edição por HDR, edição base, e edição principal
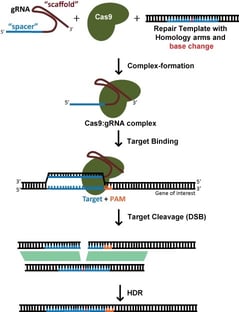
Para uma edição específica, como a inserção de uma etiqueta fluorescente ou a introdução de uma mutação específica, geralmente recorre-se à reparação dirigida pela homologia (HDR) para incorporar novas informações no ADN. Isto também requer um modelo de ADN exógeno. O HDR, contudo, é um processo muito pouco eficiente, e geralmente envolve a necessidade de clonagem de uma única célula e subsequente rastreio para edições bem sucedidas. Este é um processo muito demorado e não deve ser levado a cabo de ânimo leve! De facto, alcançar verdadeiramente o padrão de ouro requer não uma, mas duas rondas de clonagem de uma única célula – como controlo, deve-se reverter a edição de volta ao original, a fim de provar que o fenótipo se deveu realmente à edição pretendida e não a alguma variante de passageiro que veio junto com o clone de uma única célula (embora isto raramente seja feito).
Ao visar uma quebra dsDNA para HDR, a escolha do local alvo é muito mais limitada pela localização desejada da edição; a eficiência diminui drasticamente quando o local de corte é >30nt das extremidades proximais do modelo de reparação (Yang et al.., 2013). Isto significa que, para a edição de genes, existem normalmente poucos potenciais gRNAs. Embora SpCas9, com uma preferência PAM de NGG, ainda seja a enzima Cas mais utilizada, o desenvolvimento de SaCas9, NmeCas9, Cas12a enzimas, e suas variantes de engenharia, oferece opções adicionais de PAM que podem expandir muito as opções de gRNA.
Duas novas tecnologias oferecem uma alternativa ao HDR para a introdução de edições. As mesmas restrições de localização são ainda mais requintadas para o chamado editor de base Cas9, que faz alterações de DNA na ausência de quebras de dsDNA (Rees et al., 2018). Para C>T e A>G editores de base, a edição pretendida deve estar numa janela de 5 – 10 nt em relação ao PAM, e as edições espectrais são possíveis se houver outro alvo C ou A na janela. Outra tecnologia, edição principal (revista em Anzalone et al., 2020). não está limitada a transições de nucleótidos únicos mas ainda requer um PAM próximo, embora estes ainda sejam dias iniciais para esta tecnologia, e o utilizador pode necessitar de optimizar numerosos parâmetros para gerar a edição desejada.
A chave inglesa: Activação e inibição do gene por CRISPRa e CRISPRi
Finalmente, para modular a expressão do gene ao nível da transcrição – tecnologias CRISPRa (activação) e CRISPRi (inibição) – um Cas9 (dCas9) nucleo-morto é dirigido perto do promotor de um gene alvo. Aqui, a janela do alvo não é tão ampla como para o nocaute através do corte CRISPR. Para o CRISPRa, é mais eficaz visar uma janela de ~100nt a montante do local de início da transcrição (TSS), enquanto que para o CRISPRi, uma janela de ~100nt a jusante do TSS dá a maior actividade. Assim, um determinado gene terá apenas uma dúzia de gRNAs para escolher no local ideal. É também importante ter boa informação sobre a localização exacta do TSS. Diferentes bases de dados anotam o TSS de diferentes maneiras, e foi demonstrado que a base de dados FANTOM, que depende de CAGE-seq para capturar directamente o limite do mRNA, fornece o mapeamento mais preciso (Radzisheuskaya et al., 2016). Neste caso, a localização e a sequência têm aproximadamente a mesma importância na concepção – uma sequência optimizada pouco fará se estiver no local errado, mas como a janela alvo é mais de uma fila, há menos gRNA para escolher, e assim uma sequência optimizada pode não estar disponível.
Previsão da eficácia do gRNA
Nós e outros examinámos a capacidade de usar sequências e outras características para nomear gRNAs que provavelmente estarão activas, não só para SpCas9 mas também para algumas outras enzimas Cas. Parece não existir um sistema de pontuação universal para seleccionar um gRNA, uma vez que o método de produção do guia (transcrição sintética, in vitro, ou entrega lentiviral) pode afectar a precisão de uma pontuação preditiva, bem como aspectos dinâmicos do alvo (por exemplo, acessibilidade devido ao estado da cromatina). Nenhuma previsão computacional é sempre perfeita, mas isto pode diminuir o número de guias que é necessário testar em laboratório.
Importante, para qualquer modificação de interesse, seria insensato tirar conclusões com base na actividade de um único gRNA, e assim a diversidade de gRNAs através de um gene deve ser examinada sempre que possível, quando se utilizam abordagens de nocaute ou de modulação transcripcional.
Anular efeitos fora do alvo

A actividade fora do alvo dos gRNA é importante a considerar. Embora a paisagem básica de desencontros que ainda podem, no entanto, conduzir a actividade tenha sido estabelecida, e possa ser utilizada para identificar sítios susceptíveis de dar origem a um efeito fora do alvo, não há dados suficientes para prever completamente quais os sítios que irão e não irão mostrar níveis apreciáveis de modificação. A sequência de células modificadas pelo CRISPR indica que as consequências da actividade fora do alvo, pelo menos para as condições experimentais utilizadas, não levaram a mutações detectáveis (Veres et al., 2014). Ao trabalhar com clones unicelulares, os autores observam que “a heterogeneidade clonal pode representar um obstáculo mais sério à geração de linhas celulares verdadeiramente isogénicas do que os efeitos fora do alvo mediados por núcleos”. Além disso, conjuntos de dados em grande escala de centenas de ecrãs genéticos que utilizam bibliotecas de genomas têm mostrado grande concordância entre diferentes sequências que visam o mesmo gene, o que sugere que os efeitos fora do alvo não esmagaram o verdadeiro sinal nestes ensaios (Dempster et al., 2019). Mais uma vez, a estratégia experimental é clara: para qualquer gene de interesse, deve-se exigir que múltiplos gRNAs de diferentes sequências dêem origem ao mesmo fenótipo, de modo a concluir que o fenótipo se deve a um efeito on-target.
Conclusões
Selecção de gRNAs para uma experiência precisa de equilibrar a maximização da actividade on-target enquanto minimiza a actividade off-target, o que parece óbvio mas pode muitas vezes exigir decisões difíceis. Por exemplo, seria melhor utilizar um gRNA menos activo que vise um local verdadeiramente único no genoma, ou um gRNA mais activo com um local alvo adicional numa região do genoma sem função conhecida? Para a criação de modelos de células estáveis que devem ser utilizados para estudo a longo prazo, o primeiro pode ser a melhor escolha. No entanto, para que uma biblioteca de todo o genoma realize ecrãs genéticos, uma biblioteca composta pelo último seria provavelmente mais eficaz, desde que se tenha cuidado na interpretação dos resultados, exigindo que múltiplas sequências visando um gene sejam pontuadas de modo a chamar a esse gene um acerto.
Este é um momento emocionante para a genómica funcional, com uma lista sempre em expansão de ferramentas para sondar a função do gene. As melhores ferramentas são apenas tão boas como a pessoa que as utiliza, e o uso adequado da tecnologia CRISPR dependerá sempre de cuidadoso desenho experimental, execução e análise.

Muito obrigado ao nosso blogger convidado John Doench!
 John Doench é o Director do R&D na Plataforma de Perturbação Genética no Instituto Broad Institute e tem trabalhado com muitos Addgenies para ajudar a melhorar a compreensão, curadoria, e explicação dos nossos recursos CRISPR. Ele gosta realmente de pequenos RNAs.
John Doench é o Director do R&D na Plataforma de Perturbação Genética no Instituto Broad Institute e tem trabalhado com muitos Addgenies para ajudar a melhorar a compreensão, curadoria, e explicação dos nossos recursos CRISPR. Ele gosta realmente de pequenos RNAs.
p>Anzalone AV, Koblan LW, Liu DR (2020) Edição de genoma com núcleos CRISPR-Cas, editores de base, transposas e editores principais. Nat Biotechnol 38:824-844 . https://doi.org/10.1038/s41587-020-0561-9
Dempster JM, Pacini C, Pantel S, Behan FM, Green T, Krill-Burger J, Beaver CM, Younger ST, Zhivich V, Najgebauer H, Allen F, Gonçalves E, Shepherd R, Doench JG, Yusa K, Vazquez F, Parts L, Boehm JS, Golub TR, Hahn WC, Root DE, Garnett MJ, Tsherniak A, Iorio F (2019) Acordo entre dois grandes conjuntos de dados de dependência genética pan-cancer CRISPR-Cas9. Nat Commun 10: . https://doi.org/10.1038/s41467-019-13805-y
Doench JG (2017) Estou pronto para o CRISPR? Um guia do utilizador para ecrãs genéticos. Nat Rev Genet 19:67-80 . https://doi.org/10.1038/nrg.2017.97
Hanna RE, Doench JG (2020) Concepção e análise das experiências CRISPR-Cas. Nat Biotechnol 38:813-823 . https://doi.org/10.1038/s41587-020-0490-7p>Radzisheuskaya A, Shlyueva D, Müller I, Helin K (2016) A optimização da posição sgRNA melhora acentuadamente a eficiência da repressão transcripcional mediada pelo CRISPR/dCas9. Ácidos Nucleicos Res 44:e141-e141 . https://doi.org/10.1093/nar/gkw583
Rees HA, Liu DR (2018) Edição de base: química de precisão sobre o genoma e transcriptoma de células vivas. Nat Rev Genet 19:770-788 . https://doi.org/10.1038/s41576-018-0059-1
Veres A, Gosis BS, Ding Q, Collins R, Ragavendran A, Brand H, Erdin S, Cowan CA, Talkowski ME, Musunuru K (2014) Baixa Incidência de Mutações Fora do Alvo em Clones de Células-Tronco Humanas Individuais CRISPR-Cas9 e TALEN Targeted Human Stem Cell Clones Detected by Whole-Genome Sequencing. Células estaminais 15:27-30 . https://doi.org/10.1016/j.stem.2014.04.020
Yang L, Guell M, Byrne S, Yang JL, De Los Angeles A, Mali P, Aach J, Kim-Kiselak C, Briggs AW, Rios X, Huang P-Y, Daley G, Church G (2013) Optimização da edição do genoma de células estaminais humanas sem cicatrizes. Nucleic Acids Research 41:9049-9061 . https://doi.org/10.1093/nar/gkt555
Recursos no Blog Addgene
- Ouvir o nosso Podcast com John Doench
- Aprender como conduzir ecrãs de biblioteca conjunta CRISPR a nível do genoma
- Ler outras publicações do Blog CRISPR
0 comentários